多房室模型#
作者: jing-alice
多房室神经元模型旨在模拟生物大脑中神经元的真实形态,这种模型包含多个树突,结构较为复杂,计算量大,因此目前使用场景较少。通常,一个多房室神经元模型既可以单独使用,研究单个神经元的特性,也可以用多个此类模型组合成神经网络,探索神经环路的特性。一般来说,神经环路中的多房室神经元数量从几个到几千个不等。
然而,目前还缺乏一个可以支持用户自定义具有任意结构的多房室模型的框架。这会限制科研人员对复杂的神经动力学和连接模式的研究,从而阻碍神经科学领域的发展。
NeurAI 通过提供良好简单的接口,方便用户自定义多房室模型结构,为用户提供了实际可行的解决方案。
本章将从以下几个方面进行详细介绍:
单房室 主要介绍了单房室结构模型。
自定义多房室模型 主要介绍了如何使用本框架 NeurAI 创建自定义结构的多房室模型。
示例 则为用户提供一个简单的三房室模型示例,并提供了相关源码。
单房室#
通常一个神经元( Fig1. neuron)如图一所示,由三个主要部分组成:细胞体,树突和轴突,这涉及复杂的生理特性。常用的神经元模型,如泄露整合发放(LIF)模型和霍奇金-赫胥黎(HH)模型等,都只是在单个房室的简单假设下,仅考虑离子通道产生的动作电位,并没有考虑到电位沿着树突和轴突进行传播的复杂情况。相比之下,多房室模型可以更加准确和全面地表示神经元行为。
Fig1. neuron#
多房室模型通过考虑不同的房室如细胞体、树突和和轴突等,捕捉到神经元的解剖特征,使得相关科学研究人员能够探索复杂的树突处理、精细的信息传导动力学以及不同房室的特定功能。通过考虑不同房室内离子通道的多样性,多房室模型有可能启发出新性质以及研究出特定功能的形成机制。
因此,多房室模型对于深入理解突触整合、网络动力学效应以及神经计算的内在复杂性,具有重要意义。基于以上优势,虽然多房室模型的资源消耗较大,但它提供了一种全面的模型工具,可以揭示神经元功能的深层机制,推进对神经动力学和信息处理的理解。
图2(Fig2. Neuronal Discretization)展示了皮质锥体神经元的示意图,以及通过建立一系列的房室对其结构近似模拟。 在不同的多房室模型中,使用的房室数量可以从数千个到图中最右角描述的1个不等。
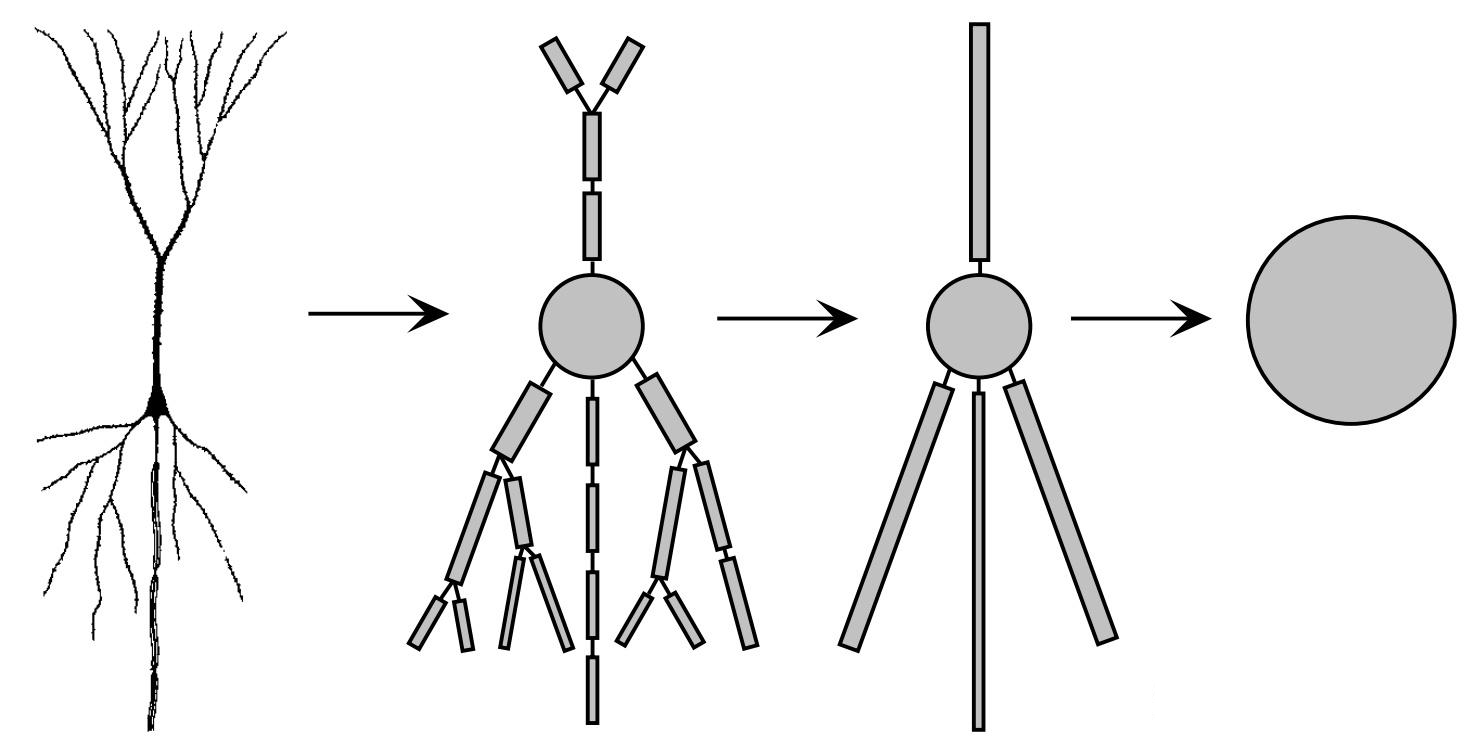
Fig2. Neuronal Discretization#
在图2(Fig2. Neuronal Discretization)中,从左到右依次为:完整的神经元、八个房室的神经元、四个房室的神经元和单个房室的神经元。 每个房室都包含一个单独的电压值。完整的神经元模型最为复杂,而单个神经元模型最为简单。
在多房室模型中,每个房室都有自己的膜电位,表示为 \(V_{m}\),以及各个房室内部的相关电流。 在多房室模型中,每个房室通常与两个相邻房室相互连接。多房室模型包括的基本元素有:
细胞体房室#
此处,\(V_{m}^{s}\) 表示细胞体的膜电压, \(C_{m}^{s}\) 表示膜电容,\(g_{L}^{s}\) 表示电导,\(E_{L}^{s}\) 表示静息电位, \(I_{a}^{i,s}\) 表示与细胞体相连的第 \(ith\) 个房室的电流,\(C^{s}\) 表示与细胞体相连的所有房室的集合, \(I_{syn}^{j,s}\) 描述来自突触前神经元对细胞体的的突触电流输入,\(S^{s}\) 表示与细胞体相连的突触前神经元的集合, \(I_{ext}^{s}\) 表示流入细胞体的外部电流。
树突房室#
树突房室的动力学遵循与细胞体相似的方程,不包含适应电流,额外添加了两个控制树突脉冲变化的电流。
此处,\(I_{Na}^{d}\) 和 \(I_{K_{dr}}^{d}\) 分别表示钠(Na+)和延迟整流钾(K+)电流。
房室间的电流#
每个房室接收一个轴向电流,该电流是所有流入该室以及来自连接房室的轴向电流之和。
此处 \(C^{k}\) 表示与 \(kth\) 房室相连的所有房室集合。每个房室特定的轴向电流由下式给出:
此处 \(g_{c}^{i,k}\) 表示 \(ith\) 房室 和 \(kth\) 房室之间的耦合电导。
突触电流#
每个房室可以接收AMPA、NMDA或GABA等突触电流,数学描述如下:
自定义多房室模型#
在 NeurAI 框架中,用户可以根据自己的特定需求灵活创建任意结构的多房室模型并进行仿真,大致分为三个步骤:定义房室结构、定义网络、仿真网络。
定义房室结构#
用户可以通过 neurai.nn.neuron.multi_compartment.compartment.LeakyIF 类来定义细胞体房室和树突房室等。
通过 neurai.nn.neuron.multi_compartment.compartment.MCConn 实现两个房室之间的连接。
所有的突触都在 neurai.nn.neuron.multi_compartment.compartment 中提供,包括AMPA,GABA和NMDA。
用户定义的多房室模型必须继承自 neurai.nn.neuron.multi_compartment.mc_neuron.MCNeuron ,在继承的类中定义任意结构的多房室模型。
from neurai.nn.neuron.multi_compartment import MCNeuron, LeakyIF, AMPA, NMDA, MCConn, MCConnSynapse, MCSynapse, ConnGen
class MCDemo(MCNeuron):
size: int = 1
def setup(self):
# Somatic compartment
self.soma = LeakyIF(size=self.size, gL=2.95, C=58.90)
# Dendritic compartments
self.apical = LeakyIF(self.size, gL=3.53, C=70.69)
self.basal = LeakyIF(self.size, gL=2.12, C=42.41)
# Synapses
self.ampa = AMPA(size=self.size, g=1, t_decay=2, V_rest=0.)
# Connections
self.connsynapse = MCConnSynapse(pre=self.ampa, post=self.apical)
self.conn = MCConn(pre=self.apical, post=self.soma, g=10)
self.synapse = MCSynapse(pre=None, post=self.ampa, conn=jnp.asarray([i + 1 for i in range(self.size)]))
定义网络#
创建网络需要继承 neurai.nn.snet.SNet ,在继承的类中构建网络模型。
from neurai.nn import SNet, SpikeGenerator
class SimNet(SNet):
def setup(self):
self.size = 35
spike_times = [50.1]
self.I = SpikeGenerator(size=self.size, spike_times=spike_times)
self.mc_neuron1 = MCDemo(self.size)
# connect to AMPA
self.connmcsynapse = ConnGen(self.I, self.mc_neuron1.synapse1)
网络模拟#
net = MultiCompartment()
param = net.init()
# parameters
sim_t = 400
dt = 0.1
xs = jnp.arange(int(sim_t / dt)) * dt
monitors_list = [MonitorConf('MCDemo_0', 'soma'), MonitorConf('MCDemo_0', 'basal'), MonitorConf('MCDemo_0', 'apical')]
monitorbs = MonitorBS(monitors=monitors_list, recorder=MemoryRecorder())
print("run")
mon = net.run(param, input=None, t=sim_t, monitor=monitorbs)
示例#
示例1:一个带有树突的基础多房室模型#
一个带有树突的基础多房室模型如图:Fig3. a_basic_compartmental_model (a) 所示,其中包含一个细胞体以及与细胞体耦合的两个树突。
为了测试该示例模型的动力学行为,给细胞体房室传入了外部电流(总共持续400 ms 的100 pA),并记录了所有房室变化的电压值,( Fig3. a_basic_compartmental_model ) (b) )。 正如预期的那样,在被施加电流的房室(细胞体房室)观察到最大值,而距离较远的房室则受到的影响较小,该模型能够成功捕捉到生物神经元信号的衰减特性。 在这个模型中,细胞体对不同数量的突触输入(5-35个突触)也会产生不同的反应,如右侧图( Fig3. a_basic_compartmental_model (c) )所示。

Fig3. a_basic_compartmental_model#
以下是 Fig3. a_basic_compartmental_model (b) 的完整示例:
import os
import sys
from neurai.config import set_platform, set_simulate_status
set_platform(platform="gpu")
set_simulate_status(enable_simulate_jit=True, multi_thread_connect=True)
from neurai.nn.neuron.multi_compartment import MCNeuron, LeakyIF, AMPA, NMDA, MCConn, MCConnSynapse
from neurai.nn import Module, SNetLayer, SNet
from neurai.monitor import MonitorBS, MonitorConf, MemoryRecorder
from neurai.util import visualization
import jax.numpy as jnp
import time
from jax import config
config.update("jax_enable_x64", True)
time_start = time.time()
class MCDemo(MCNeuron):
size: int = 1
def setup(self):
self.soma = LeakyIF(size=self.size, gL=2.95, C=58.90)
self.apical = LeakyIF(self.size, gL=3.53, C=70.69)
self.basal = LeakyIF(self.size, gL=2.12, C=42.41)
self.ampa = AMPA(size=self.size, g=1, t_decay=2, V_rest=0.)
self.nmda = NMDA(size=self.size, g=1, t_decay=60, V_rest=0.)
self.connsynapse1 = MCConnSynapse(pre=self.ampa, post=self.apical)
self.connsynapse2 = MCConnSynapse(pre=self.nmda, post=self.apical)
self.conn1 = MCConn(pre=self.apical, post=self.soma, g=10)
self.conn2 = MCConn(pre=self.basal, post=self.soma, g=10)
class SimNet(SNet):
def setup(self):
self.size = 1
self.mc_neuron1 = MCDemo(self.size)
class MultiCompartment(Module):
def setup(self):
self.network = SNetLayer(SimNet)
def __call__(self, input=None, t=0, monitor=None):
param, mon = self.network(input, t=t, monitor=monitor)
return mon
net = MultiCompartment()
print("init")
param = net.init()
time_init = time.time()
# parameters
sim_t = 700
dt = 0.1
num_compartment = 5
i_input = jnp.zeros((int(sim_t / dt), num_compartment))
# input -> soma
i_input = i_input.at[slice(1002, 5002), 0].set(100.0)
monitors_list = [MonitorConf('MCDemo_0', 'soma'), MonitorConf('MCDemo_0', 'basal'), MonitorConf('MCDemo_0', 'apical')]
monitorbs = MonitorBS(monitors=monitors_list, recorder=MemoryRecorder())
print("run")
mon = net.run(param, input=i_input, t=sim_t, monitor=monitorbs)
time_end = time.time()
v_soma = mon['MCDemo_0.soma']
v_apical = mon['MCDemo_0.apical']
v_basal = mon['MCDemo_0.basal']
if mon:
visualization.line_plot(
mon["ts"],
jnp.transpose(jnp.asarray([mon['MCDemo_0.soma'][:, 0], mon['MCDemo_0.basal'][:, 0], mon['MCDemo_0.apical'][:, 0]])),
xlabel="Time(ms)",
ylabel="Voltage(mV)",
show=True,
save=True,
title="Input \u2192 soma")
print('\nTimes:\n' + ' Total time: {:.3f} s\n'.format(time_end - time_start) +
' Time to initialize: {:.3f} s\n'.format(time_init - time_start) +
' Time to simulate: {:.3f} s\n'.format(time_end - time_init))
以下是 Fig3. a_basic_compartmental_model (c) 的完整示例:
import os
import sys
from neurai.config import set_platform, set_simulate_status
set_platform(platform="cpu")
set_simulate_status(enable_simulate_jit=True, multi_thread_connect=True)
from neurai.nn.neuron.multi_compartment import MCNeuron, LeakyIF, AMPA, NMDA, MCConn, MCConnSynapse, MCSynapse, ConnGen
from neurai.nn.neuron.spike_generator import SpikeGenerator
from neurai.nn import Module, SNetLayer, SNet
from neurai.monitor import MonitorBS, MonitorConf, MemoryRecorder
import jax.numpy as jnp
import time
from jax import config
config.update("jax_enable_x64", True)
time_start = time.time()
class MCDemo(MCNeuron):
size: int = 1
def setup(self):
self.soma = LeakyIF(size=self.size, gL=2.95, C=58.90)
self.apical = LeakyIF(self.size, gL=3.53, C=70.69)
self.basal = LeakyIF(self.size, gL=2.12, C=42.41)
self.ampa = AMPA(size=self.size, g=1, t_decay=2, V_rest=0.)
self.nmda = NMDA(size=self.size, g=1, t_decay=60, V_rest=0.)
self.connsynapse1 = MCConnSynapse(pre=self.ampa, post=self.apical)
self.connsynapse2 = MCConnSynapse(pre=self.nmda, post=self.apical)
self.conn1 = MCConn(pre=self.apical, post=self.soma, g=10)
self.conn2 = MCConn(pre=self.basal, post=self.soma, g=10)
# # j, i -> postsynaptic, presynaptic indices respectively, connect AMPA
self.synapse1 = MCSynapse(pre=None, post=self.ampa, conn=jnp.asarray([i + 1 for i in range(self.size)]))
# # j, i -> postsynaptic, presynaptic indices respectively, connect NMDA
self.synapse2 = MCSynapse(pre=None, post=self.nmda, conn=jnp.asarray([i + 1 for i in range(self.size)]))
class SimNet(SNet):
def setup(self):
self.size = 35
spike_times = [50.1]
self.I = SpikeGenerator(size=self.size, spike_times=spike_times)
self.mc_neuron1 = MCDemo(self.size)
# connect to AMPA and NMDA
self.connmcsynapse1 = ConnGen(self.I, self.mc_neuron1.synapse1)
self.connmcsynapse2 = ConnGen(self.I, self.mc_neuron1.synapse2)
class MultiCompartment(Module):
def setup(self):
self.network = SNetLayer(SimNet)
def __call__(self, input=None, t=0, monitor=None):
_, mon = self.network(input, t=t, monitor=monitor)
return mon
net = MultiCompartment()
print("init")
param = net.init()
time_init = time.time()
# parameters
sim_t = 400
dt = 0.1
xs = jnp.arange(int(sim_t / dt)) * dt
monitors_list = [MonitorConf('MCDemo_0', 'soma'), MonitorConf('MCDemo_0', 'basal'), MonitorConf('MCDemo_0', 'apical')]
monitorbs = MonitorBS(monitors=monitors_list, recorder=MemoryRecorder())
print("run")
mon = net.run(param, input=None, t=sim_t, monitor=monitorbs)
time_end = time.time()
v_soma = mon['MCDemo_0.soma']
v_apical = mon['MCDemo_0.apical']
v_basal = mon['MCDemo_0.basal']
v_soma = jnp.asarray(v_soma)
if mon:
import matplotlib.pyplot as plt
plt.figure()
for i in range(35):
if (i + 1) % 5 == 0:
plt.plot(xs, v_soma[1:, i])
plt.ylim(-75, -40)
plt.title("AMPA & NMDA")
plt.xlabel("Time (ms)")
plt.ylabel("Voltage (mV)")
plt.savefig("base_mc_input_synpatic_demo")
print('\nTimes:\n' + ' Total time: {:.3f} s\n'.format(time_end - time_start) +
' Time to initialize: {:.3f} s\n'.format(time_init - time_start) +
' Time to simulate: {:.3f} s\n'.format(time_end - time_init))
